2023年2月22日,國際學術期刊Nucleic Acids Research刊登了華中農業大學動物遺傳育種團隊開發的基因組育種大數據計算新工具HIBLUP,中文名為“天權”。論文題為“HIBLUP: An integration of statistical models on the BLUP framework for efficient genetic evaluation using big genomic data”,被期刊評為“突破性進展”論文(Breakthrough Article)。該研究系統分析了已有遺傳評估算法特點,針對現有算法在處理快速增長的基因組育種大數據時面臨的瓶頸問題,首創基于V矩陣的“HE+PCG”策略,可完全避免遺傳評估計算過程中的大矩陣求逆,開發出更適合基因組育種大數據時代的高性能計算新工具HIBLUP。與現有工具相比,HIBLUP計算速度最快且消耗內存最少,而且基因分型個體在群體中占比越大,優勢愈明顯。此外,HIBLUP軟件功能豐富、操作便捷,可運行于Windows、Linux、macOS等平臺,并且全面適配國產Kunpeng(鯤鵬)生態。
圖1.HIBLUP論文入選NAR期刊“突破性進展”論文(breakthrough article)
遺傳評估是育種的基礎,隨著基因組育種時代的來臨,育種數據規模快速增長,評估算法的計算速度已成為育種中的關鍵限制因素。遺傳評估主要包括方差組分估計及育種值求解兩個步驟,其中方差組分估計的計算復雜度高,通常數月或一年更新一次;育種值求解復雜度相對較低,需要日常計算更新。目前,國際現有育種工具(如丹麥的DMU、美國的BLUPF90、英國的ASReml等)采用的評估算法都是以混合模型方程組(Mixed model equation, MME)為核心,即MME策略,需要求解個體關系矩陣和MME左手項(Left hand side, LHS)的逆矩陣(如圖2所示)。傳統育種利用系譜構建個體親緣關系矩陣,評估過程涉及的矩陣極其稀疏,FSPAK算法(美國專利)能夠以極快速度求解稀疏矩陣LHS的逆矩陣,是基于系譜信息的傳統育種計算必不可少的核心程序。然而,隨著基因組育種時代的來臨,個體親緣關系矩陣構建逐漸由系譜過渡到基因組信息,關系矩陣及LHS矩陣也相應由全稀疏轉變為半稠密或全稠密,FSPAK算法并不適用于稠密矩陣運算,其劣勢逐漸顯現,雖然FSPAK團隊針對性地做出了優化,例如,推出了能夠自動鑒別稀疏及稠密塊的FSPAK升級版“YAMS”,以及利用區分核心群和非核心群的方式近似求解基因組個體關系逆矩陣的“APY”策略等,一定程度上提升了數據處理能力,但仍然依賴MME框架,無法避免多次大矩陣的求逆運算,當基因分型個體規模累計到數十萬時,MME策略面臨計算效率低及內存需求大的雙重問題,并不能適應基因組大數據時代的育種計算需求。為解決這一難題,HIBLUP首創基于方差協方差V矩陣的“HE+PCG”策略,即利用HE回歸法估計方差組分,采用基于V矩陣的PCG迭代法估計育種值(如圖2所示),可完全避免遺傳評估計算過程中的大矩陣求逆,并且V矩陣的維度(有表型個體數)遠低于MME方程的維度(所有個體數×遺傳隨機效應個數)。因此,無論是計算效率還是內存需求上, HIBLUP全面優于基于MME策略的現有工具,更適合基因組育種時代的大數據計算。
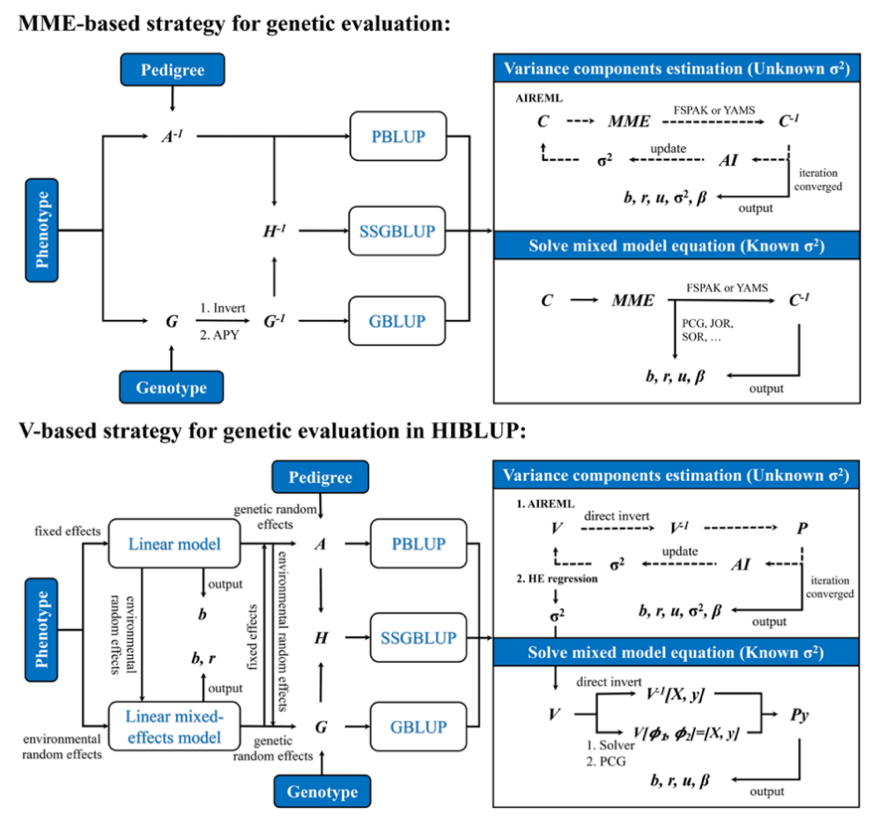
圖2.HIBLUP計算策略與國外現有工具MME計算策略的比較。針對基因組大數據的計算特點,HIBLUP首創基于V矩陣的“HE+PCG”策略,可完全避免遺傳評估計算過程中的大矩陣求逆,且V矩陣的維度相比于MME方程更小,尤其適用于多隨機效應模型,在計算效率和內存需求上, HIBLUP全面優于現有育種計算工具。
HIBLUP針對不同平臺鏈接了華為KML、Intel MKL、OpenBLAS等高性能矩陣數學計算庫,結合OpenMP等多種并行技術提升計算效率,同時運用內存映射、單雙精度混合運算等技術,大大降低計算過程中的內存消耗。如圖3所示,與國際知名育種工具相比,HIBLUP在個體親緣關系矩陣構建、單性狀及多性狀模型擬合上,均具有明顯的優勢,計算速度最快,內存消耗最少。通過模擬UKB級別大數據(50萬個體、100萬標記)進行測試發現,HIBLUP采用的“HE+PCG”策略能夠在1小時完成方差組分估計及育種值求解,其他軟件需要長達數周甚至數月的時間。此外,通過模擬不同表型個體數以及不同基因型個體占比的多種組合方式,對比不同軟件擬合SSGBLUP模型的效率時發現,基因分型個體在群體中占比越大,HIBLUP的計算性能優勢愈明顯。
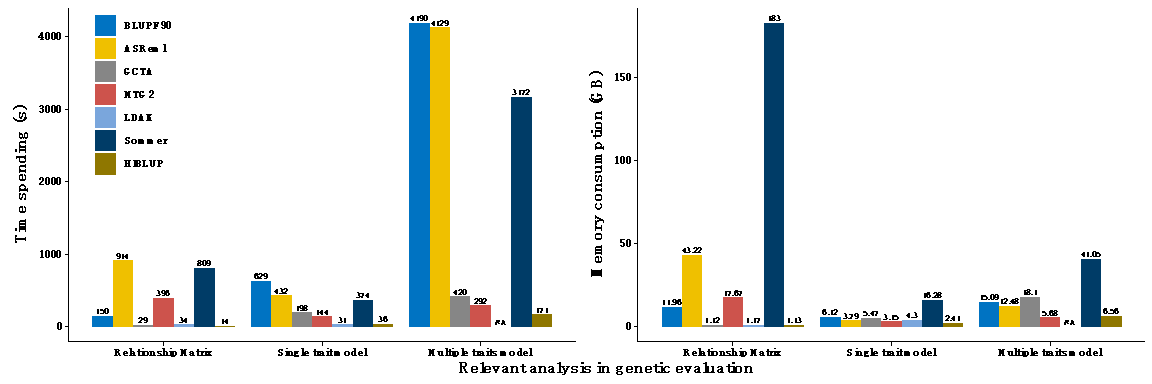
圖3.HIBLUP 新工具與國外現有工具計算時間及內存消耗對比。測試數據集的群體大小為10000,多性狀模型中性狀個數為2,均采用32線程并行運算,統一采用GBLUP模型。NA表示對應軟件未實現該功能模塊。
HIBLUP軟件自2018年初全國畜牧總站組織基因組育種算法交流時啟動研發,歷經5年,在功能模塊、計算性能、用戶體驗等方面不斷升級完善,目前已被來自全球50多個國家的用戶使用。HIBLUP具備豐富的遺傳分析功能,包含常用的單性狀模型、重復記錄模型、多性狀模型等,支持環境互作、遺傳互作、環境與遺傳互作等分析,是目前唯一兼具基因組選種及基因組精準選配功能的育種計算工具。HIBLUP不僅可運行于Windows、Linux、Mac OS等國外平臺,而且全面適配國產華為Kunpeng(鯤鵬)生態。目前,HIBLUP已在揚翔、中糧、海大、金旭等多個大型農牧企業應用,為我國種豬基因組高效選育,以及三元商品豬生產精準選配提供了國產化新工具。
華中農業大學博士后尹立林和武漢理工大學博士生張浩浩為論文共同第一作者,華中農業大學趙書紅教授、劉小磊教授和李新云教授為論文共同通訊作者。該研究受到國家重點研發計劃青年科學家項目、國家自然科學基金、國家生豬體系崗位科學家項目的資助。
HIBLUP軟件下載及使用教程網站見:https://www.hiblup.com
原文鏈接:https://doi.org/10.1093/nar/gkad074